摘要:本试验以果蝇(Drosophilamelanogaster)ATP合成酶16kDa蛋白脂质C亚基基因(vha16)cDNA序列为信息探针,对蜜蜂EST数据库进行同源检索筛选,克隆了蜜蜂ATP合成酶16kDa蛋白脂质C亚基基因(vha16)的cDNA全序列(GenBank登记号为AY343324),该基因全长581bp。经RT-PCR克隆、序列分析验证,结果表明与电子克隆序列完全一致。该基因具有完整的开放阅读框(ORF),编码蛋白为156个氨基酸。通过设计特异引物,克隆到长为2968bp的该基因的部分基因组序列。利用GeneFinder软件分析发现,在蜜蜂vha16基因组序列的第1~133位、第1965~2154位和第2711~2968位分别为外显子;2个内含子分别位于第134~1964位和第2155~2710位,且内含子的剪切位置均符合GT-AG规则。
关键词:蜜蜂(Apismellifera);电子克隆;TR-PCR;ATP合成酶16kDa蛋白脂质C亚基基因(vha16);基因结构
基于表达序列标签(expressedsequencetags,ESTs)的电子克隆(insilicocloning)策略,是近年来发展起来的一门快速克隆基因的新技术,其技术核心是利用生物信息学技术组装延伸ESTs序列,获得基因的部分乃至全长cDNA序列进一步利用RT-PCR的方法进行克隆分析、验证。随着EST数据库的进一步完善,电子克隆策略已成为克隆新基因的重要方法,并成功地应用于人类基因组的研究[1-3]。在过去的几年间,在许多动物的质膜上都发现了运输质子的液泡型ATP合成酶(vacuolar-typeATPase,V-ATPase)[4-7]。在昆虫的生理系统中,V-ATPase起着至关重要的作用,它可以产生穿膜电压,为次级主动运输过程如K+/2H+逆向、氨基酸/K+同向跨越质膜运输提供能量[8-9]。而蜜蜂(Apismellifera)V-ATPase基因序列尚未见报道。
本研究采用电子克隆的方法,以果蝇(Drosophilamelanogaster)ATP合成酶16kDa蛋白脂质C亚基基因(vha16)cDNA序列为信息探针,BLAST检索GenBank的蜜蜂EST数据库,拼接有部分同源的EST序列,获得蜜蜂vha16基因的cDNA全序列并经RT-PCR验证。并在该基因的cDNA序列的基础上,设计系列特异引物,进一步克隆了蜜蜂vha16基因组部分序列,并分析该基因组序列的结构。
1材料与方法
1.1材料
1.1.1载体、菌株及蜜蜂
克隆载体pMD-T为TaKaRa公司产品;宿主菌E.coliTG1为本实验室保存。试验所用蜜蜂由福建农林大学蜂学学院教学蜂场提供。
1.1.2工具酶及试剂
T4DNA连接酶、plusTaqDNA聚合酶、蛋白酶K及各种限制性内切酶均为TaKaRa公司产品;逆转录试剂盒为MBI产品;DNAMarker购自上海生工生物工程公司。
1.2方法
1.2.1总RNA的提取及反转录合成单链cDNA[10]
取0.5g成年工蜂的头部组织,加入10mlTrizol冰浴研磨,然后加入终浓度为0.3g·L-1的糖原,用1ml注射器反复吸打。室温放置5min后加入2ml氯仿,剧烈混合3min,11000r/min离心15min。上清移至无菌且RNasefree的离心管中,加入2倍体积乙醇,12000r/min离心5min;弃上清,以75%乙醇洗涤沉淀2次。稍干后,用无RNase的灭菌水溶解沉淀。cDNA的合成按反转录试剂盒说明书步骤进行。
1.2.2蜜蜂基因组DNA的提取
取成年工蜂10只,参照LIFTOND的方法[11]提取蜜蜂基因组DNA,干燥后溶于100μLTE,-20℃保存备用。
1.2.3引物的设计与合成
根据电子克隆所得的完整cDNA序列,设计合成特异引物(由上海生工生物工程公司合成):
引物1:F:5’-CGACAACCTGTAAATACTCGTG-3’
R:5’-GGTTGCAGGCGACGAGTCTTGC-3’
引物2:F:5’-CGACAACCTGTAAATACTCGTG-3’
R:5’-TGTACCTGACTTTGCTGTGCCG-3’
引物3:F:5’-GTTCTAATTGCTGGTGGTCTAG-3’
R:5’-GGTTGCAGGCGACGAGTCTTGC-3’
1.2.4PCR扩增蜜蜂vha16基因片段
用所设计的引物1,以供试蜜蜂cDNA为模板扩增特异片段。PCR程序为94℃预变性2min,94℃变性30s,54℃复性45s,72℃延伸1min。共30个循环,72℃延伸10min。反应完毕后取10μl于15g·L-1琼脂糖凝胶进行电泳分析。
1.2.5蜜蜂vha16基因组序列克隆
分别用所设计的特异引物2、3,以供试蜜蜂基因组DNA为模板,用plusTaq酶扩增特异片段。PCR程序为94℃预变性5min,94℃变性1min,56℃复性45s,72℃延伸90s。共35个循环,72℃延伸10min。反应完毕后取10μL于12g·L-1琼脂糖凝胶进行电泳分析。
1.2.6重组、克隆和DNA序列分析
用低熔点琼脂糖凝胶电泳回收PCR产物,与pMD-T载体(摩尔比3:1)连接,挑选阳性克隆,委托TaKaRa公司进行DNA序列测定。
1.2.7蜜蜂vha16基因组序列内含子/外显子分析
利用GeneFinder软件(http://www.bioscience.org/urllists/genefind.htm)进行基因组序列的内含子和外显子分析。将蜜蜂vha16基因的cDNA序列和相应的基因组序列进行对齐显示,以确定该基因的外显子和内含子结构。部分区域根据典型的剪切位点(GT-AG法则)进行调整。
2结果与分析
2.1蜜蜂vha16基因cDNA序列的电子克隆
从http://www.ncbi.nlm.nih.gov/的GenBank的核酸(nr)数据库中检索昆虫vha16基因,获得果蝇的vha16基因cDNA序列NM_05745[12],以该序列为模板对蜜蜂EST数据库进行BLAST检索,获得与之部分同源的蜜蜂EST群,从中选取一条EST(BI513093)作为种子序列BLAST检索蜜蜂EST数据库,将检出与种子序列同源性较高或有部分重叠的EST序列拼接组装为重叠群(contig),再以此重叠群序列重复以上BLAST检索过程,反复进行EST重叠群序列的拼接和比对,直至检出所有的重叠EST或重叠群不能继续延伸[13],最终获得581bp的蜜蜂vha16基因的cDNA全序列,由蜜蜂dbEST中的BI513093、BI513122、BI514568、BI511976、BI508009、BI505241和BI511110等EST序列拼接而成(图1);经RESearch软件分析,发现其具有完整的开放式阅读框架(ORF),起始密码子在第59位,终止密码子在第527位,推测编码156个氨基酸。
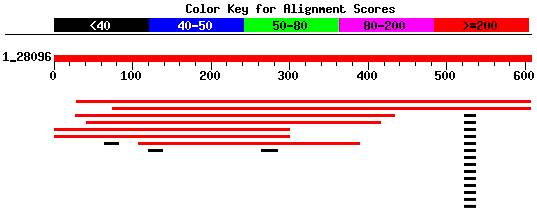
图1蜜蜂vha16cDNA序列在蜜蜂EST数据库中的比对结果
2.2RT-PCR验证
PCR结果经15g·L-1琼脂糖凝胶电泳,结果出现单一条带,约570bp,与预计大小相符(图2)。用pMD-T载体克隆后测序,结果与电子克隆序列一致(图3)。蜜蜂vha16基因的完整序列的GenBank登记号为AY343324。
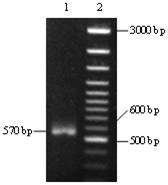
图2蜜蜂vha16cDNART-PCR产物的琼脂糖凝胶电泳分析鉴定
1:RT-PCR产物;2:DNA标准分子量

2.3蜜蜂vha16基因组序列克隆
分别用所设计的特异引物2、3扩增特异片段,PCR结果经12g·L-1琼脂糖凝胶电泳,结果出现单一条带,分别约为2126bp和978bp(图未列)。用pMD-T载体克隆后测序所得的2个片段序列,有146bp的重叠区域,结合电子克隆序列,可拼接获得长为2968bp序列(篇幅所限,序列未列出),但cDNA两端序列未能得到延伸。
2.4基因结构分析
利用GeneFinder软件分析基因组序列的内含子和外显子发现,对蜜蜂vha16基因组序列分析发现,在其第1~133位、第1965~2154位和第2711~2968位分别为外显子;2个内含子分别位于第134~1964位和第2155~2710位,且内含子的剪切位置均符合GT-AG规则。
3讨论
利用EST资料的电子克隆是克隆功能基因的新途径,与传统的基因克隆方法相比,具有快捷、成本低、针对性强等特点,但也受到dbEST的EST数量和质量的限制[14],因此在EST资料非常丰富的模式物种(如人、鼠等)中应用较多。在实际应用中,以模式物种某一已知基因序列为起点,结合目标物种EST数据库,采用现代生物信息学及实验验证相结合的技术方法,尤其是通过同源性比较完全有可能快速筛选到一些具有重要功能的基因。
同时,通过蛋白同源比较也可看出,蜜蜂vha16基因的推测蛋白与已知昆虫VHA16的大小相似。所以,笔者认为本实验中电子克隆的蜜蜂vha16基因cDNA序列是完整的,至少可以说该基因的cDNA序列,推测编码的蛋白序列是完整的,具有完整的ORF。在该基因的结构分析研究中,曾试图扩增非转录区序列,但未能取得成功,这对于全面分析该基因的结构有一定的影响。
参考文献
[1]WeiMh,KaravanovaI,IvanovSV,etal.Insilico-initiatedcloningandmolecularcharacterizationofanovelhumanmemberoftheL1genefamilyofneuralcelladhesionmolecules.HumanGenetics,1998,103(3):355-364
[2]ZhangC,YuY,ZhangS,etal.Characterization,chromosomalassignment,andtissueexpressionofanovelhumangenebelongingtotheARFGAPfamily.Genomics,2000,63(3):400-408
[3]WestAB,LockhartPJ,O'farellC,FarrerMJ.Identificationofanovelgenelinkedtoparkinviaabi-directionalpromoter.JournalofMolecularBiology,2003,326(1):11-19
[4]FinbowME,GoodwinSF,MeagherL,etal.Evidencethatthe16kDaproteolipid(subunitc)ofthevacuolarH+-ATPaseandductinfromgapjunctionsarethesamepolypeptideinDrosophilaandManduca:molecularcloningoftheVha16kgenefromDrosophila.JournalofCellScience,1994,107:1817-1824
[5]AzumaM,OhtaY.ChangesinH+-translocatingvacuolar-typeATPaseintheanteriorsilkglandcellofBombyxmoriduringmetamorphosis.TheJournalofExperimentalBiology,1998,201:479-486
[6]WieczorekH,GruberG,HarveyWR,etal.TheplasmamembraneH+-V-ATPasefromtobaccohornwormmidgut.JournalofBioenergeticsandBiomembranes,1999,31:67-74
[7]VitavskaO,WieczorekH,MerzendorferH.AnovelroleforsubunitCinmediatingbindingoftheH+-V-ATPasetotheactincytoskeleton.TheJournalofBiologicalChemistry,2003,278:18499-18505
[8]LepierA,AzumaM,HarveyWR,WieczorekH.K+/H+antiportinthetobaccohornwormmidgut:theK+-transportingcomponentoftheK+pump.TheJournalofExperimentalBiology,1994,196:361-373
[9]AzumaM,HarveyWR,WieczorekH.StoichiometryofK+/H+antiporthelpstoexplainextracellularpH11inamodelepithelium.FEBSLetters,1995,361:153-156
[10]ChomczynskiP,SachiN.Single-stepmethodofRNAisolationbyAcidguanidiumthiocyanate-phenol-chloroformextraction.AnalyticalBiochemistry,1987,162:156-159
[11]夏庆友.RAPD的扩增条件、重复性及遗传模型研究.蚕业科学,1996,22(1):20-25
[12]AdamsMD,CelnikerSE,HoltRA,etal.ThegenomesequenceofDrosophilamelanogaster.Science,2000,287:2185-2195
[13]张德礼,孙晓静,凌伦奖,陈润生,马大龙.人类SR蛋白超家族新成员——SFRS12(SRrp508)的基因克隆和特性分析.遗传学报,2002,29(5):377-383湖北天马养蜂场,加我们的微信一起学养蜂。
[14]PrigentC,GillR,TrowerM,SanseauP.Insilicocloningofanewproteinkinase,Aik2,relatedtoDrosophilaaurorausingthenewtool:ESTBlast.InsilicoBiology,1999,1(2):123-128
陈大福福建农林大学蜂学学院